数据准备 :使用 sample 函数对原始数据进行随机抽样,抽取 70% 的数据作为训练集(n=329),其余的作为测试集(n=141),抽样结果(训练集)保存在 sample.txt 文件中。
建立模型 :使用 stats 程序包中的 glm 函数对训练集数据使用单因素和多因素 Logistic 回归分析,确定发生植体周炎的危险因素(P < 0.05)。用多因素 Logistic 回归分析的结果计算 OR 值,并借助 boot 程序包,使用 Bootstrap 法计算 OR 的 95% 置信区间。使用 rms 程序包建立列线图预测模型。
验证模型 :分别借助 rms 程序包中的 calibrate 函数和 pROC 程序包中的 plot.roc 函数,使用 C-index 和校准曲线来检验模型的区分度和校准度。Logistic 回归的 C-index 即为 ROC 曲线下面积(AUC)。内部验证和外部验证分别使用训练集和测试集数据进行。
导入需要用到的 R 程序包并导入数据:
1
2
3
4
5
6
7
8
9
# 导入 R 库
library ( readxl )
library ( writexl )
# library(lme4)
library ( broom )
library ( tidyverse )
# 导入数据
raw_data <- read_excel ( "data.xlsx" )
根据需要预处理数据 1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
# 选择研究变量
data <- raw_data[ , c ( -1 , -2 , -3 , -10 , -17 , -18 , -20 ) ]
data $ 年龄 <- raw_data $ 修复体戴入日期 - raw_data $ 出生日期
# 计数数据
names.count <- c ( "吸烟" , "性别" , "修复体材料" , "骨吸收情况" , "分期" , "分级" ,
"颌位" , "牙位" , "种植系统" , "植骨手术明细" , "用膜明细" , "缝合方式" )
names.all <- colnames ( data ) [ - which ( colnames ( data ) == "植体周炎" ) ]
# 再次处理数据细节
data $ 口内植体数目 <- as.numeric ( data $ 口内植体数目) # “口内植体数目” 原本是字符串,这里将其转换为数值
data [which ( data $ 分级 == "B" ), ] $ 分级 <- "b" # 分级 B 和 b 相同
data [which ( data $ 分级 == "a" ), ] $ 分级 <- "b" # 把分级里a合并到b里面
data [which ( data $ 骨吸收情况 == "1/2" ), ] $ 骨吸收情况 <- ">1/2"
data [which ( as.numeric ( data $ 修复体材料) >= 3 ), ] $ 修复体材料 <- 3 # 修复体材料 3 以后都合并为一类
# 处理错误数据
data [which ( data $ 吸烟 == 2 ), ] $ 吸烟 <- 0
data [which ( is.na ( data $ 种植系统)), ] $ 种植系统 <- 1
data [which ( is.na ( data $ 用膜明细)), ] $ 用膜明细 <- 1
# 去除有缺失值的病例
data <- data [complete.cases ( data ), ]
# 计数数据转成因子型变量
for ( name in names.count ) {
data[[name]] <- factor ( data[[name]] )
}
现在准备好的数据储存在 data 变量中。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
#> # A tibble: 6 × 23
#> 吸烟 种植前空腹血糖 性别 修复体材料 口内植体数目 骨吸收情况 分期 分级
#> <fct> <dbl> <fct> <fct> <dbl> <fct> <fct> <fct>
#> 1 0 4.9 1 1 4 <1/3 3 b
#> 2 0 4.9 1 1 4 1/3-1/2 3 b
#> 3 0 5.98 1 1 3 <1/3 3 c
#> 4 0 4.89 2 1 7 1/3-1/2 3 c
#> 5 0 4.89 2 1 7 植体 3 c
#> 6 0 4.89 2 1 7 1/3-1/2 4 c
#> # ℹ 15 more variables: 颌位 <fct>, 牙位 <fct>, `角化组织宽度(mm)` <dbl>,
#> # `维护周期(月)` <dbl>, 平均复查周期 <dbl>, 种植系统 <fct>, 植体直径 <dbl>,
#> # 植体长度 <dbl>, 植骨手术明细 <fct>, 用膜明细 <fct>, 缝合方式 <fct>,
#> # `穿龈角度(近中)` <dbl>, `穿龈角度(远中)` <dbl>, 植体周炎 <dbl>,
#> # 年龄 <drtn>
通过以下代码随机选取 7/10 的数据作为训练集,并将抽样方案保存在 sample.txt 文件中。
1
2
smp <- sample ( seq_len ( nrow ( data )), round ( nrow ( data ) * 0.7 ))
write ( smp , file = "sample.txt" )
代码编写过程中可能需要多次运行代码,为保持多次运行之间的一致性,在选取合适的抽样(单因素 Logistic 回归 )后,之后的抽样选取就通过读取 sample.txt 文件完成。
1
2
3
4
# 随机抽取训练集,其他作为测试集
smp <- scan ( file = "sample.txt" )
data.train <- data[smp , ]
data.test <- data[ - smp , ]
使用 glm 函数,对训练集数据的每个预测变量进行单因素分析,获取 P 值,并选出 P < 0.05 的预测变量(抽样的不同会对这一步造成较大影响)。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
p.values <- data.frame ()
names.selected <- c ()
for ( name in names.all ) {
f <- as.formula ( paste ( c ( "植体周炎" , paste ( c ( "`" , name , "`" ), collapse = "" )), collapse = "~" ))
model.tmp <- glm ( f , data = data.train , family = binomial ())
additions <- summary ( model.tmp ) $ coefficients[ -1 , 4 ]
if ( is.null ( names ( additions ))) {
names ( additions ) <- paste ( name , levels ( data.train[[name]] ) [ -1 ] , sep = "" )
}
p.values <- rbind ( p.values , data.frame ( "P value" = additions ))
if ( any ( additions < 0.05 )) {
names.selected <- c ( names.selected , name )
}
}
names.selected
1
2
#> [1] "修复体材料" "骨吸收情况" "分级" "维护周期(月)"
#> [5] "平均复查周期"
根据单因素 Logistic 回归的结果,这次抽样选出的危险因素(P < 0.05)为:“修复体材料”,“骨吸收情况”,“分级”,“维护周期(月)”和“平均复查周期”。
用单因素 Logistic 回归筛选出的危险因素,同样使用 glm 函数,进行多因素 Logistic 回归分析。
1
2
3
4
5
6
7
8
9
names.05 <- names.selected
f.05 <- as.formula ( paste ( collapse = "~" ,
c (
"植体周炎" ,
paste ( collapse = "" , c ( "`" , paste ( names.05 , collapse = "`+`" ), "`" ))
)
))
model.05 <- glm ( f.05 , data = data.train , family = binomial ())
summary ( model.05 )
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
#>
#> Call:
#> glm(formula = f.05, family = binomial(), data = data.train)
#>
#> Coefficients:
#> Estimate Std. Error z value Pr(>|z|)
#> (Intercept) -5.26989 0.72909 -7.23 4.9e-13 ***
#> 修复体材料2 -0.07740 0.41125 -0.19 0.85072
#> 修复体材料3 0.95364 0.52022 1.83 0.06678 .
#> 骨吸收情况>1/2 2.81191 0.63639 4.42 9.9e-06 ***
#> 骨吸收情况1/3-1/2 0.66028 0.44389 1.49 0.13689
#> 骨吸收情况植体 1.12159 0.52408 2.14 0.03235 *
#> 分级c 1.08745 0.44902 2.42 0.01544 *
#> `维护周期(月)` 0.02031 0.00576 3.52 0.00042 ***
#> 平均复查周期 0.01974 0.00941 2.10 0.03593 *
#> ---
#> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#>
#> (Dispersion parameter for binomial family taken to be 1)
#>
#> Null deviance: 283.80 on 328 degrees of freedom
#> Residual deviance: 219.58 on 320 degrees of freedom
#> AIC: 237.6
#>
#> Number of Fisher Scoring iterations: 6
注意这里构建公式的方法:先使用加了 collapse 参数的 paste 函数将 names.05 拼接成字符串形式的“公式”,再通过 as.formula 方法,将字符串转换为公式。
在 R 中,可以使用 exp(coef(model)) 计算比值比(OR)。
既可以借助 boot 程序包,使用 Bootstrap 法 计算 OR 及其 95% 置信区间,也可以直接使用confint 函数
为了控制更多的细节,这里使用 boot 程序包的 boot 函数计算 OR,并使用同属于 boot 程序包的 boot.ci 函数计算置信区间。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
library ( boot )
set.seed ( 1645248416 ) # unclass(Sys.time())
# 计算 OR 值的函数
st.boot <- function ( data , indices ) {
model <- glm ( f.05 , data[indices , ] , family = binomial ())
return ( exp ( coef ( model )))
}
# 运行 Bootstrap 计算 OR
results <- boot ( data.train , statistic = st.boot , R = 1000 )
ci <- matrix ( nrow = length ( results $ t0 ), ncol = 2 )
colnames ( ci ) <- c ( "2.5%" , "97.5%" )
rownames ( ci ) <- names ( results $ t0 )
for ( i in seq_len ( length ( results $ t0 ))) {
n <- boot.ci ( boot.out = results , conf = 0.95 , type = "bca" , index = i )
ci[i , ] <- n $ bca [c ( 4 , 5 ) ]
}
table2.05 <- data.frame (
"OR" = results $ t0 ,
"OR.CI" = ci ,
"P值" = tidy ( model.05 ) $ p.value
)
head ( table2.05 )
1
2
3
4
5
6
7
#> OR OR.CI.2.5. OR.CI.97.5. P值
#> (Intercept) 0.005144 0.001077 0.02295 4.901e-13
#> 修复体材料2 0.925524 0.365046 2.13859 8.507e-01
#> 修复体材料3 2.595143 0.856581 7.63863 6.678e-02
#> 骨吸收情况>1/2 16.641733 4.311440 62.61887 9.938e-06
#> 骨吸收情况1/3-1/2 1.935325 0.718117 4.88898 1.369e-01
#> 骨吸收情况植体 3.069725 0.762045 8.27774 3.235e-02
1
2
3
4
5
6
table_simple <- data.frame (
"OR" = exp ( coef ( model.05 )),
"OR.CI" = exp ( confint ( model.05 )),
"P值" = tidy ( model.05 ) $ p.value
)
head ( table_simple )
1
2
3
4
5
6
7
#> OR OR.CI.2.5.. OR.CI.97.5.. P值
#> (Intercept) 0.005144 0.001112 0.01965 4.901e-13
#> 修复体材料2 0.925524 0.403301 2.04682 8.507e-01
#> 修复体材料3 2.595143 0.913069 7.12754 6.678e-02
#> 骨吸收情况>1/2 16.641733 4.901419 60.79632 9.938e-06
#> 骨吸收情况1/3-1/2 1.935325 0.811607 4.67088 1.369e-01
#> 骨吸收情况植体 3.069725 1.063397 8.47141 3.235e-02
OR 和 P 值与之前的结果相同,但置信区间有少许差别。
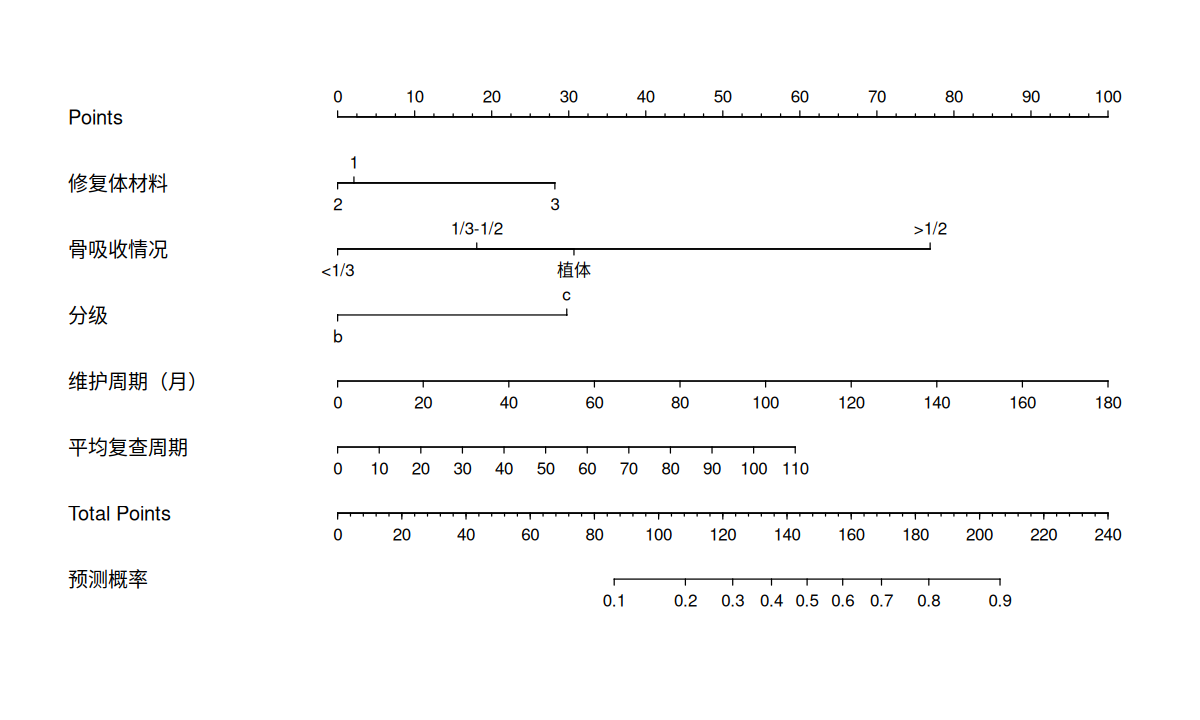
rms 程序包提供了用来建立列线图预测模型的函数。
1
2
3
4
5
6
7
library ( rms )
# 使用训练集建立列线图预测模型
fit.train <- lrm ( f.05 , data.train , x = TRUE , y = TRUE )
ddist.train <- datadist ( data.train )
options ( datadist = ddist.train )
plot ( nomogram ( fit.train , fun = plogis , lp = FALSE , funlabel = "预测概率" ))
数据验证分为内部验证和外部验证。这里使用测试集进行外部验证,内部验证在训练集上进行。在内部验证和外部验证中,都分别通过 ROC 曲线 和列线图校准曲线 检验数据的区分度和校准度。
特异度和敏感度
由FeanDoe - Modified version from Walber's Precision and Recall <a class="external free" href="https://commons.wikimedia.org/wiki/File:Precisionrecall.svg">https://commons.wikimedia.org/wiki/File:Precisionrecall.svg</a>,CC BY-SA 4.0 ,链接
ROC 曲线全称受试者工作特征曲线 (receiver operating characteristic curve),又称为感受性曲线 (sensitivity curve),曲线上每个点反映着对同一信号刺激的感受性。
ROC 曲线的横轴为特异度 (specificity),即真阴性率 (true negative rate,TNR);纵轴为敏感度 (sensitivity),即真阳性率 (true postive rate,TPR)、召回率 (Recall)。
AUC 全称曲线下面积 (Area Under Curve),在 Logistic 回归中,AUC 等同于 C-index(concordance index,一致性指数),可以直观的评价模型的好坏,值越大越好。
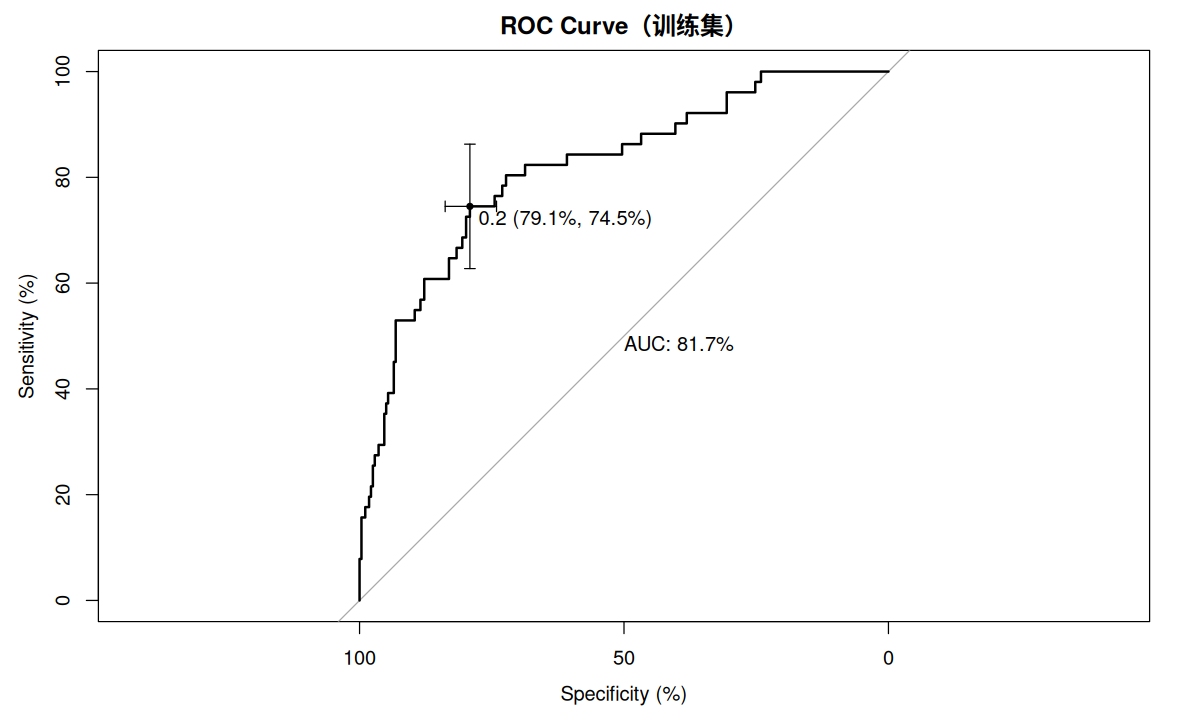
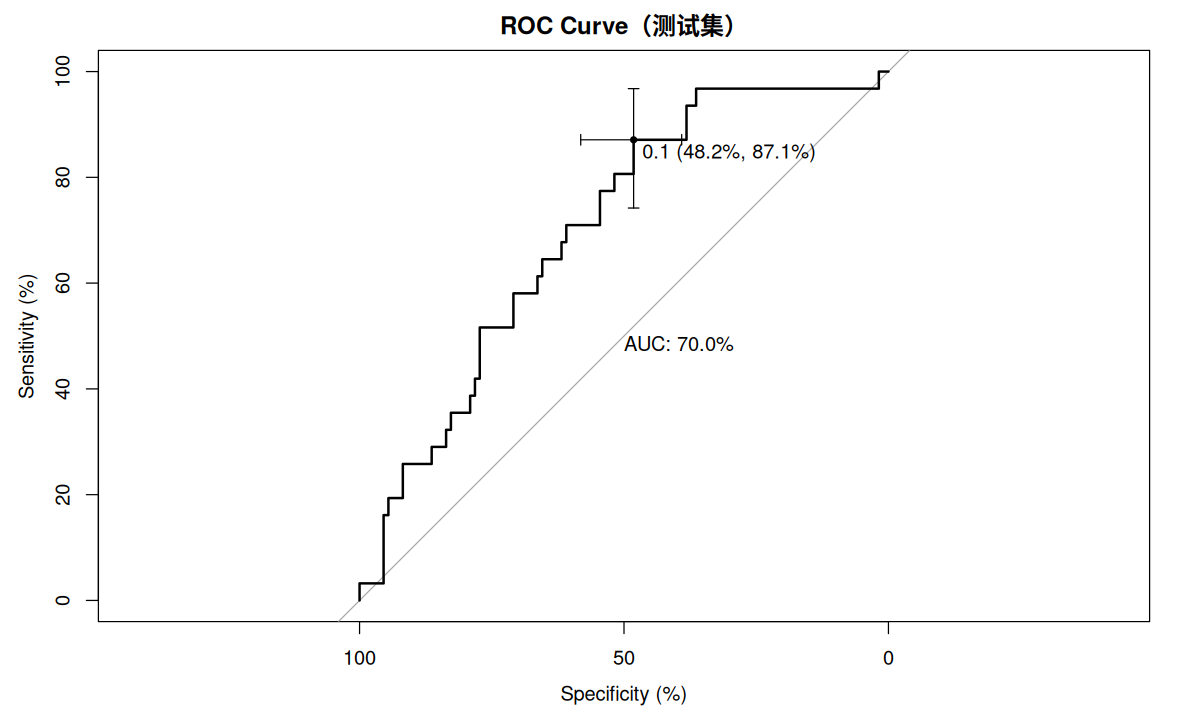
使用 ROC 曲线检验数据的区分度,pROC 程序包提供了这里所需要的函数,其中 plot.roc 用于绘制 ROC 曲线,ci.auc 用于获取 AUC 的置信区间。
1
2
3
4
5
6
7
8
pre.train <- predict ( model.05 , data.train , type = c ( "response" ))
p.roc.train <- plot.roc ( data.train $ 植体周炎, pre.train ,
main = "ROC Curve(训练集)" , percent = TRUE ,
print.auc = TRUE ,
ci = TRUE , of = "thresholds" ,
thresholds = "best" ,
print.thres = "best" )
ci.auc ( p.roc.train )
1
#> 95% CI: 75.2%-88.2% (DeLong)
1
2
3
4
5
6
7
8
pre.test <- predict ( model.05 , data.test , type = c ( "response" ))
p.roc.test <- plot.roc ( data.test $ 植体周炎, pre.test ,
main = "ROC Curve(测试集)" , percent = TRUE ,
print.auc = TRUE ,
ci = TRUE , of = "thresholds" ,
thresholds = "best" ,
print.thres = "best" )
ci.auc ( p.roc.test )
1
#> 95% CI: 60.2%-79.8% (DeLong)
训练集 AUC 达 81.7%(95%CI:75.16%-88.25%),测试集 AUC 为 70.0%(95%CI:60.24%-79.76%),提示列线图模型区分度良好。
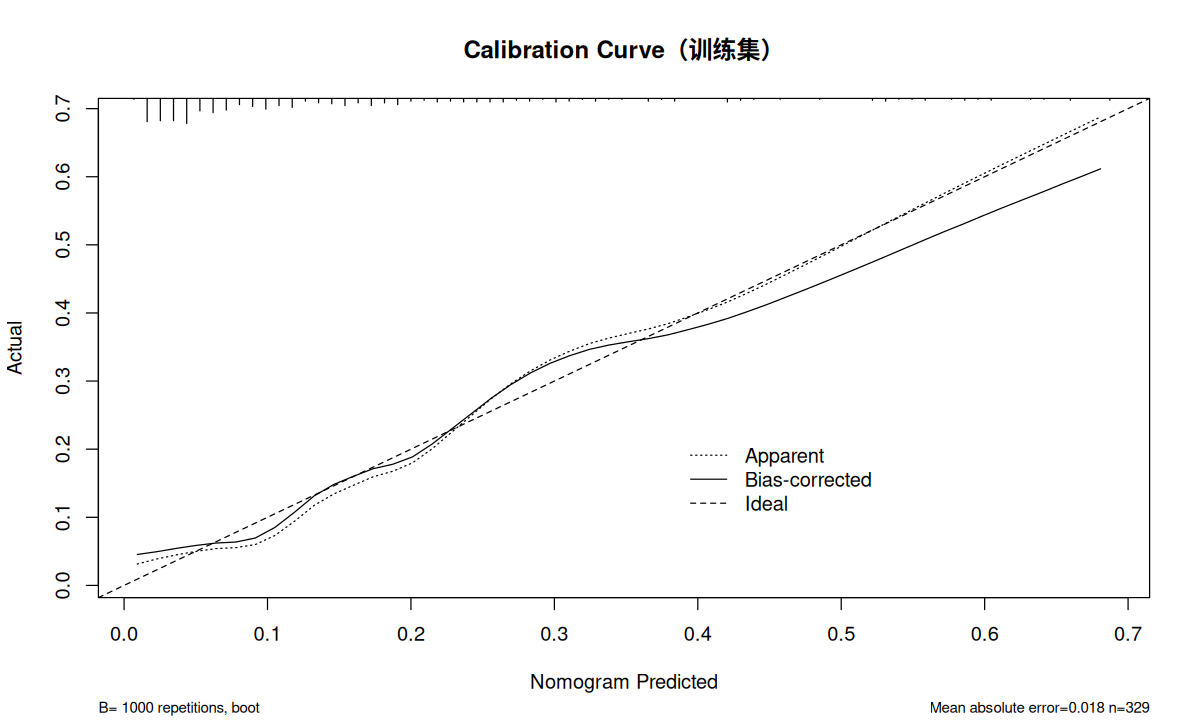
使用列线图校准曲线检验数据的校准度。绘制列线图校准曲线用到的 calibrate 也是 rms 包中的函数。这里使用 Bootstrap 法(method = "boot"),重复 1000 次(B = 1000)。其他常用的 method 还有 "crossvalidation"、".632" 及 "randomization"。
1
2
3
4
5
cal_train <- calibrate ( fit.train , method = "boot" , B = 1000 )
plot ( cal_train ,
xlab = "Nomogram Predicted" ,
ylab = "Actual" ,
main = "Calibration Curve(训练集)" )
1
2
3
4
5
6
7
8
9
fit.test <- lrm ( f.05 , data.test , x = TRUE , y = TRUE )
ddist.test <- datadist ( data.test )
options ( datadist = ddist.test )
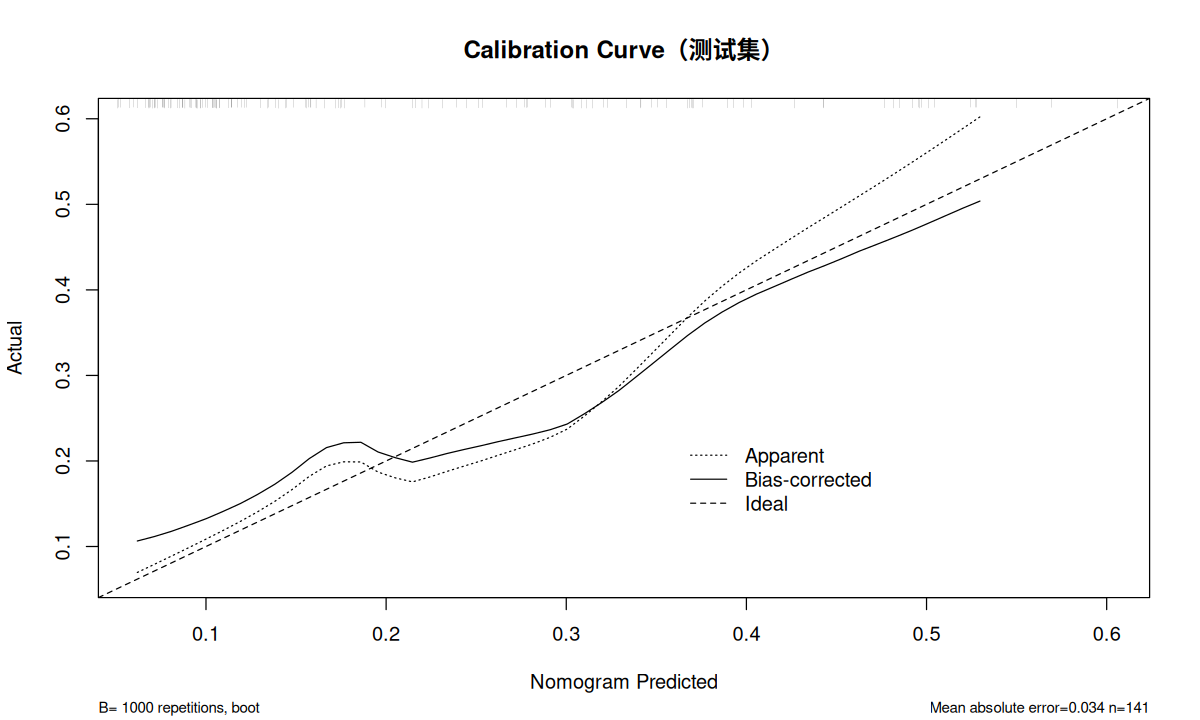
cal_test <- calibrate ( fit.test , method = "boot" , B = 1000 )
plot ( cal_test ,
xlab = "Nomogram Predicted" ,
ylab = "Actual" ,
main = "Calibration Curve(测试集)" )
通过校验曲线可以看出训练集和测试集的预测值与实际值基本一致。